fit#
- scipy.stats.fit(dist, data, bounds=None, *, guess=None, method='mle', optimizer=<function differential_evolution>)[source]#
将离散或连续分布拟合到数据
给定分布、数据和分布参数的界限,返回参数的最大似然估计值。
- 参数:
- dist
scipy.stats.rv_continuous或scipy.stats.rv_discrete 表示要拟合到数据的分布的对象。
- data一维 array_like
要将分布拟合到的数据。 如果数据包含任何
np.nan、np.inf或 -np.inf,则 fit 方法将引发ValueError。- boundsdict 或元组序列,可选
如果是一个字典,则每个键是分布的参数名称,对应的值是一个包含该参数下限和上限的元组。如果分布仅为该参数的有限值范围定义,则不需要该参数的条目;例如,某些分布的参数必须位于区间 [0, 1] 上。参数 location (
loc) 和 scale (scale) 的界限是可选的;默认情况下,它们分别固定为 0 和 1。如果是一个序列,则元素 i 是一个包含分布的第 i 个参数的下限和上限的元组。在这种情况下,必须提供所有分布形状参数的界限。可选地,location 和 scale 的界限可以跟在分布形状参数之后。
如果要固定形状(例如,如果已知),则下限和上限可以相等。如果用户提供的下限或上限超出定义分布的域的范围,则分布域的界限将替换用户提供的值。类似地,必须为整数的参数将被约束为用户提供的界限内的整数值。
- guessdict 或 array_like,可选
如果是一个字典,则每个键是分布的参数名称,对应的值是该参数值的猜测值。
如果是一个序列,则元素 i 是分布的第 i 个参数的猜测值。在这种情况下,必须提供所有分布形状参数的猜测值。
如果未提供 guess,则决策变量的猜测值将不会传递给优化器。 如果提供了 guess,则任何缺失参数的猜测值将设置为下限和上限的平均值。 必须为整数的参数的猜测值将四舍五入为整数值,并且位于用户提供的界限和分布域的交集之外的猜测值将被裁剪。
- method{‘mle’, ‘mse’}
使用
method="mle"(默认),通过最小化负对数似然函数来计算拟合。 对于超出分布支持的观察结果,应用大的、有限的惩罚(而不是无限的负对数似然)。 使用method="mse",通过最小化负对数乘积间距函数来计算拟合。 对于超出支持的观察结果,应用相同的惩罚。 我们遵循 [1] 的方法,该方法已针对重复观察的样本进行了推广。- optimizer可调用对象,可选
optimizer 是一个可调用对象,它接受以下位置参数。
- fun可调用对象
要优化的目标函数。 fun 接受一个参数
x,即分布的候选形状参数,并返回给定x、dist 和提供的 data 的目标函数值。 optimizer 的工作是找到最小化 fun 的决策变量的值。
optimizer 还必须接受以下关键字参数。
- bounds元组序列
决策变量值的界限;每个元素将是一个包含决策变量的下限和上限的元组。
如果提供了 guess,则 optimizer 还必须接受以下关键字参数。
- x0array_like
每个决策变量的猜测值。
如果分布有任何必须为整数的形状参数,或者如果分布是离散的并且位置参数未固定,则 optimizer 还必须接受以下关键字参数。
- integrality布尔值 array_like
对于每个决策变量,如果决策变量必须约束为整数值,则为 True,如果决策变量是连续的,则为 False。
optimizer 必须返回一个对象,例如
scipy.optimize.OptimizeResult的一个实例,该对象在属性x中保存决策变量的最佳值。 如果提供了属性fun、status或message,则它们将包含在fit返回的结果对象中。
- dist
- 返回:
- result
FitResult 具有以下字段的对象。
- paramsnamedtuple
一个 namedtuple,其中包含分布的形状参数、位置和(如果适用)比例的最大似然估计值。
- successbool 或 None
优化器是否认为优化成功终止。
- messagestr 或 None
优化器提供的任何状态消息。
该对象具有以下方法
- nllf(params=None, data=None)
默认情况下,给定 data 的拟合 params 处的负对数似然函数。 接受一个包含分布的替代形状、位置和比例的元组以及替代数据的数组。
- plot(ax=None)
将拟合分布的 PDF/PMF 叠加在数据的归一化直方图上。
- result
另请参阅
备注
当用户提供包含最大似然估计值的紧密界限时,优化更有可能收敛到最大似然估计值。 例如,当将二项分布拟合到数据时,每个样本的基础实验次数可能是已知的,在这种情况下,可以固定相应的形状参数
n。参考文献
[1]Shao, Yongzhao, and Marjorie G. Hahn. “Maximum product of spacings method: a unified formulation with illustration of strong consistency.” Illinois Journal of Mathematics 43.3 (1999): 489-499.
示例
假设我们希望将分布拟合到以下数据。
>>> import numpy as np >>> from scipy import stats >>> rng = np.random.default_rng() >>> dist = stats.nbinom >>> shapes = (5, 0.5) >>> data = dist.rvs(*shapes, size=1000, random_state=rng)
假设我们不知道数据是如何生成的,但我们怀疑它遵循具有参数 n 和 p 的负二项分布。(参见
scipy.stats.nbinom。)我们认为参数 n 小于 30,并且我们知道参数 p 必须位于区间 [0, 1] 上。我们将此信息记录在变量 bounds 中,并将此信息传递给fit。>>> bounds = [(0, 30), (0, 1)] >>> res = stats.fit(dist, data, bounds)
fit在用户指定的 bounds 中搜索与数据最佳匹配的值(从最大似然估计的角度来看)。 在这种情况下,它找到了与实际生成数据的形状值相似的值。>>> res.params FitParams(n=5.0, p=0.5028157644634368, loc=0.0) # may vary
我们可以通过将分布的概率质量函数(具有拟合到数据的形状)叠加在数据的归一化直方图上来可视化结果。
>>> import matplotlib.pyplot as plt # matplotlib must be installed to plot >>> res.plot() >>> plt.show()
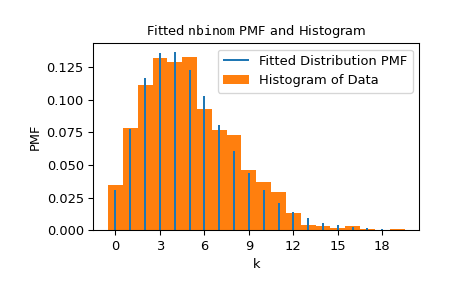
请注意,n 的估计值正好是整数;这是因为
nbinomPMF 的域仅包含整数 n,并且nbinom对象“知道”这一点。nbinom也知道形状 p 必须是 0 到 1 之间的值。 在这种情况下 - 当分布相对于参数的域是有限的时 - 我们不需要指定参数的界限。>>> bounds = {'n': (0, 30)} # omit parameter p using a `dict` >>> res2 = stats.fit(dist, data, bounds) >>> res2.params FitParams(n=5.0, p=0.5016492009232932, loc=0.0) # may vary
如果我们希望强制分布以固定为 6 的 n 进行拟合,我们可以将 n 的下限和上限都设置为 6。 但是请注意,在这种情况下,通常要优化的目标函数的值会更差(更高)。
>>> bounds = {'n': (6, 6)} # fix parameter `n` >>> res3 = stats.fit(dist, data, bounds) >>> res3.params FitParams(n=6.0, p=0.5486556076755706, loc=0.0) # may vary >>> res3.nllf() > res.nllf() True # may vary
请注意,前面示例的数值结果是典型的,但它们可能会有所不同,因为
fit使用的默认优化器scipy.optimize.differential_evolution是随机的。 但是,我们可以自定义优化器使用的设置以确保可重复性 - 甚至可以使用完全不同的优化器 - 使用 optimizer 参数。>>> from scipy.optimize import differential_evolution >>> rng = np.random.default_rng() >>> def optimizer(fun, bounds, *, integrality): ... return differential_evolution(fun, bounds, strategy='best2bin', ... rng=rng, integrality=integrality) >>> bounds = [(0, 30), (0, 1)] >>> res4 = stats.fit(dist, data, bounds, optimizer=optimizer) >>> res4.params FitParams(n=5.0, p=0.5015183149259951, loc=0.0)