gaussian_kde#
- class scipy.stats.gaussian_kde(dataset, bw_method=None, weights=None)[source]#
使用高斯核的核密度估计的表示。
核密度估计是一种以非参数方式估计随机变量的概率密度函数 (PDF) 的方法。
gaussian_kde适用于单变量和多变量数据。 它包括自动带宽确定。 对于单峰分布,估计效果最佳;双峰或多峰分布往往过度平滑。- 参数:
- datasetarray_like
从中估计的数据点。 在单变量数据的情况下,这是一个一维数组,否则是一个二维数组,形状为(#维度,#数据)。
- bw_methodstr,标量或可调用对象,可选
用于计算带宽因子的方法。 这可以是“scott”、“silverman”、标量常量或可调用对象。 如果是标量,则将直接用作 factor。 如果是可调用对象,它应该以
gaussian_kde实例作为唯一参数并返回一个标量。 如果为 None(默认),则使用“scott”。 有关更多详细信息,请参见 Notes。- weightsarray_like,可选
数据点的权重。 这必须与数据集的形状相同。 如果为 None(默认),则假定样本具有相同的权重
- 属性:
- datasetndarray
gaussian_kde初始化时使用的数据集。- dint
维数。
- nint
数据点的数量。
- neffint
有效数据点的数量。
1.2.0 版本新增。
- factorfloat
从
covariance_factor获得的带宽因子。- covariancendarray
核协方差矩阵; 这是数据协方差矩阵乘以带宽因子的平方,例如
np.cov(dataset) * factor**2。- inv_covndarray
covariance 的逆。
方法
evaluate(points)在一组点上评估估计的 pdf。
__call__(points)在一组点上评估估计的 pdf。
integrate_gaussian(mean, cov)将估计的密度乘以多元高斯分布,并在整个空间上积分。
integrate_box_1d(low, high)计算两个边界之间的一维 pdf 的积分。
integrate_box(low_bounds, high_bounds[, ...])计算矩形区间上 pdf 的积分。
integrate_kde(other)计算此核密度估计与另一个核密度估计的乘积的积分。
pdf(x)在提供的一组点上评估估计的 pdf。
logpdf(x)在提供的一组点上评估估计的 pdf 的对数。
resample([size, seed])从估计的 pdf 中随机采样一个数据集。
set_bandwidth([bw_method])使用给定的方法计算带宽因子。
计算带宽因子 factor。
注释
带宽选择强烈影响从 KDE 获得的估计值(比内核的实际形状影响更大)。 带宽选择可以通过“经验法则”、交叉验证、“插件方法”或其他方式完成; 请参阅 [3], [4] 进行评论。
gaussian_kde使用经验法则,默认设置为 Scott 规则。Scott 规则 [1], 实现为
scotts_factor, 是n**(-1./(d+4)),
其中
n是数据点的数量,d是维数。 在非等权重点的情况下,scotts_factor变为neff**(-1./(d+4)),
其中
neff是有效数据点的数量。 Silverman 对多元数据的建议 [2], 实现为silverman_factor, 是(n * (d + 2) / 4.)**(-1. / (d + 4)).
或者在非等权重点的情况下
(neff * (d + 2) / 4.)**(-1. / (d + 4)).
请注意,这与“Silverman 的经验法则” [6] 不同,后者在单变量情况下可能更稳健; 请参阅
set_bandwidth方法的文档,以实现自定义带宽规则。有关核密度估计的良好一般描述可以在 [1] 和 [2] 中找到,此多维实现的数学原理可以在 [1] 中找到。
对于一组加权样本,有效数据点的数量
neff定义为neff = sum(weights)^2 / sum(weights^2)
如 [5] 中所述。
gaussian_kde当前不支持位于其表达空间的较低维度子空间中的数据。 对于此类数据,请考虑执行主成分分析/降维,并将gaussian_kde用于转换后的数据。参考文献
[3]B.A. Turlach, “核密度估计中的带宽选择:回顾”,CORE 和 Institut de Statistique,第 19 卷,第 1-33 页,1993 年。
[4]D.M. Bashtannyk 和 R.J. Hyndman, “用于内核条件密度估计的带宽选择”,计算统计与数据分析,第 36 卷,第 279-298 页,2001 年。
[5]Gray P. G., 1969, Journal of the Royal Statistical Society. Series A (General), 132, 272
[6]核密度估计。 维基百科。 https://en.wikipedia.org/wiki/Kernel_density_estimation
示例
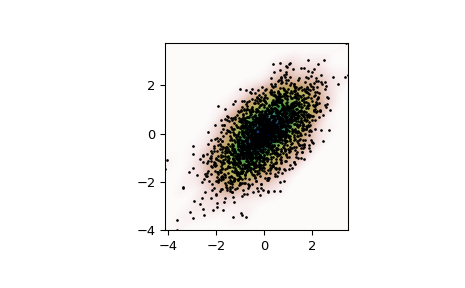
生成一些随机的二维数据
>>> import numpy as np >>> from scipy import stats >>> def measure(n): ... "Measurement model, return two coupled measurements." ... m1 = np.random.normal(size=n) ... m2 = np.random.normal(scale=0.5, size=n) ... return m1+m2, m1-m2
>>> m1, m2 = measure(2000) >>> xmin = m1.min() >>> xmax = m1.max() >>> ymin = m2.min() >>> ymax = m2.max()
对数据执行核密度估计
>>> X, Y = np.mgrid[xmin:xmax:100j, ymin:ymax:100j] >>> positions = np.vstack([X.ravel(), Y.ravel()]) >>> values = np.vstack([m1, m2]) >>> kernel = stats.gaussian_kde(values) >>> Z = np.reshape(kernel(positions).T, X.shape)
绘制结果
>>> import matplotlib.pyplot as plt >>> fig, ax = plt.subplots() >>> ax.imshow(np.rot90(Z), cmap=plt.cm.gist_earth_r, ... extent=[xmin, xmax, ymin, ymax]) >>> ax.plot(m1, m2, 'k.', markersize=2) >>> ax.set_xlim([xmin, xmax]) >>> ax.set_ylim([ymin, ymax]) >>> plt.show()
与某个点的手动 KDE 进行比较
>>> point = [1, 2] >>> mean = values.T >>> cov = kernel.factor**2 * np.cov(values) >>> X = stats.multivariate_normal(cov=cov) >>> res = kernel.pdf(point) >>> ref = X.pdf(point - mean).sum() / len(mean) >>> np.allclose(res, ref) True