NumericalInversePolynomial#
- class scipy.stats.sampling.NumericalInversePolynomial(dist, *, mode=None, center=None, domain=None, order=5, u_resolution=1e-10, random_state=None)#
基于多项式插值的CDF反演(PINV)。
PINV是数值反演的一种变体,其中使用牛顿插值公式逼近逆CDF。区间
[0,1]被分割成几个子区间。在每个子区间中,逆CDF在节点(CDF(x),x)处针对子区间中的某些点x构建。如果给定了PDF,则使用具有 5 个点的自适应 Gauss-Lobatto 积分,从给定的 PDF 数值计算 CDF。子区间会被分割,直到达到请求的精度目标。该方法不是精确的,因为它只产生近似分布的随机变量。 然而,最大容忍的近似误差可以设置为分辨率(当然,受机器精度的限制)。我们使用 u 误差
|U - CDF(X)|来测量误差,其中X是对应于分位数U的近似百分位数,即X = approx_ppf(U)。我们称最大容忍的 u 误差为算法的 u 分辨率。可以同时选择插值多项式的阶数和 u 分辨率。请注意,非常小的 u 分辨率值是可能的,但会增加设置步骤的成本。
必须在设置步骤中计算插值多项式。然而,它只适用于具有有界域的分布;对于具有无界域的分布,尾部被截断,使得尾部区域的概率与给定的 u 分辨率相比很小。
只有当PDF是单峰的或当PDF在两个模式之间不消失时,插值多项式的构造才有效。
对于给定的分布有一些限制
分布的支持(即 PDF 严格为正的区域)必须是连通的。 实际上,这意味着 PDF “不太小”的区域必须是连通的。 单峰密度满足此条件。 如果违反此条件,则可能会截断分布的域。
当 PDF 以数值方式积分时,给定的 PDF 必须是连续的并且应该是平滑的。
PDF 必须是有界的。
当分布具有重尾(因为逆CDF在0或1处变得非常陡峭)并且请求的 u 分辨率非常小时,该算法会出现问题。 例如,当请求的 u 分辨率小于 1.e-12 时,柯西分布很可能会显示此问题。
- 参数:
- distobject
具有
pdf或logpdf方法的类的实例,可以选择具有cdf方法。pdf: 分布的PDF。 PDF的签名应为:def pdf(self, x: float) -> float,即PDF应接受Python浮点数并返回Python浮点数。它不需要积分为1,即PDF不需要归一化。此方法是可选的,但需要指定pdf或logpdf之一。如果两者都给出,则使用logpdf。logpdf: 分布的PDF的对数。签名与pdf的签名相同。 同样,PDF的归一化常数的对数可以忽略。此方法是可选的,但需要指定pdf或logpdf之一。如果两者都给出,则使用logpdf。cdf: 分布的CDF。 此方法是可选的。如果提供,则可以计算“u-error”。请参阅u_error。必须与 PDF 具有相同的签名。
- modefloat, optional
分布的(精确)模式。 默认为
None。- centerfloat, optional
分布的模式或平均值的近似位置。 此位置提供有关PDF主要部分的一些信息,并用于避免数值问题。 默认为
None。- domainlist or tuple of length 2, optional
分布的支持。 默认为
None。 当None如果分布对象 dist 提供了
support方法,则它用于设置分布的域。否则,假定支持为 \((-\infty, \infty)\)。
- orderint, optional
插值多项式的阶数。 有效的阶数介于 3 和 17 之间。阶数越高,近似值的间隔越少。 默认为 5。
- u_resolutionfloat, optional
设置最大容忍的 u-error。 u_resolution 的值必须至少为 1.e-15,最多为 1.e-5。 请注意,大多数均匀随机数源的分辨率为 2-32= 2.3e-10。 因此,1.e-10 的值会导致一种可以称为精确的反演算法。 对于大多数模拟,最大误差的稍微大一点的值也足够了。 默认为 1e-10。
- random_state{None, int,
numpy.random.Generator, numpy.random.RandomState}, optional用于生成均匀随机数流的底层 NumPy 随机数生成器或种子。 如果 random_state 为 None(或 np.random),则使用
numpy.random.RandomState单例。 如果 random_state 是一个 int,则使用一个新的RandomState实例,并以 random_state 为种子。 如果 random_state 已经是Generator或RandomState实例,则使用该实例。
- 属性:
intervals获取计算中使用的间隔数。
方法
cdf(x)给定分布的近似累积分布函数。
ppf(u)给定分布的近似PPF。
qrvs([size, d, qmc_engine])给定RV的准随机变量。
rvs([size, random_state])从分布中抽样。
set_random_state([random_state])设置底层均匀随机数生成器。
u_error([sample_size])使用蒙特卡罗模拟估计近似值的 u 误差。
参考文献
[1]Derflinger, Gerhard, Wolfgang Hörmann, and Josef Leydold. “Random variate generation by numerical inversion when only the density is known.” ACM Transactions on Modeling and Computer Simulation (TOMACS) 20.4 (2010): 1-25.
[2]UNU.RAN 参考手册,第 5.3.12 节,“PINV - 基于多项式插值的CDF反演”,https://statmath.wu.ac.at/software/unuran/doc/unuran.html#PINV
例子
>>> from scipy.stats.sampling import NumericalInversePolynomial >>> from scipy.stats import norm >>> import numpy as np
要创建一个从标准正态分布中抽样的生成器,请执行以下操作
>>> class StandardNormal: ... def pdf(self, x): ... return np.exp(-0.5 * x*x) ... >>> dist = StandardNormal() >>> urng = np.random.default_rng() >>> rng = NumericalInversePolynomial(dist, random_state=urng)
创建生成器后,可以通过调用
rvs方法从分布中抽取样本>>> rng.rvs() -1.5244996276336318
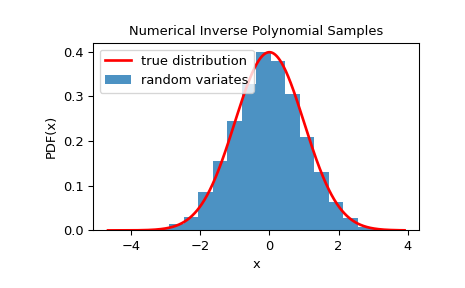
为了检查随机变量是否紧密跟随给定的分布,我们可以查看它的直方图
>>> import matplotlib.pyplot as plt >>> rvs = rng.rvs(10000) >>> x = np.linspace(rvs.min()-0.1, rvs.max()+0.1, 1000) >>> fx = norm.pdf(x) >>> plt.plot(x, fx, 'r-', lw=2, label='true distribution') >>> plt.hist(rvs, bins=20, density=True, alpha=0.8, label='random variates') >>> plt.xlabel('x') >>> plt.ylabel('PDF(x)') >>> plt.title('Numerical Inverse Polynomial Samples') >>> plt.legend() >>> plt.show()
如果在设置期间可以使用精确的CDF,则可以估计近似PPF的 u 误差。 为此,在初始化期间传递一个具有分布的精确CDF的 dist 对象
>>> from scipy.special import ndtr >>> class StandardNormal: ... def pdf(self, x): ... return np.exp(-0.5 * x*x) ... def cdf(self, x): ... return ndtr(x) ... >>> dist = StandardNormal() >>> urng = np.random.default_rng() >>> rng = NumericalInversePolynomial(dist, random_state=urng)
现在,可以通过调用
u_error方法来估计 u 误差。 它运行蒙特卡罗模拟来估计 u 误差。 默认情况下,使用 100000 个样本。 要更改此设置,可以将样本数作为参数传递>>> rng.u_error(sample_size=1000000) # uses one million samples UError(max_error=8.785994154436594e-11, mean_absolute_error=2.930890027826552e-11)
这将返回一个 namedtuple,其中包含最大 u 误差和平均绝对 u 误差。
可以通过降低 u 分辨率(最大允许的 u 误差)来降低 u 误差
>>> urng = np.random.default_rng() >>> rng = NumericalInversePolynomial(dist, u_resolution=1.e-12, random_state=urng) >>> rng.u_error(sample_size=1000000) UError(max_error=9.07496300328603e-13, mean_absolute_error=3.5255644517257716e-13)
请注意,这会增加设置时间。
可以通过调用
ppf方法来评估近似PPF>>> rng.ppf(0.975) 1.9599639857012559 >>> norm.ppf(0.975) 1.959963984540054
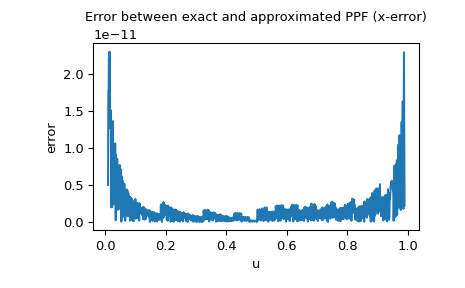
由于正态分布的PPF可以作为特殊函数使用,因此我们还可以检查 x 误差,即近似PPF和精确PPF之间的差异
>>> import matplotlib.pyplot as plt >>> u = np.linspace(0.01, 0.99, 1000) >>> approxppf = rng.ppf(u) >>> exactppf = norm.ppf(u) >>> error = np.abs(exactppf - approxppf) >>> plt.plot(u, error) >>> plt.xlabel('u') >>> plt.ylabel('error') >>> plt.title('Error between exact and approximated PPF (x-error)') >>> plt.show()