简单均匀比率 (SROU)#
必需:PDF,PDF 下的面积(如果与 1 不同)
可选:模式,模式下的 CDF
速度
设置:快
采样:慢
SROU 基于均匀比率方法,该方法使用通用不等式来构造(通用)边界矩形。它适用于具有 T(x) = -1/sqrt(x) 的 T 凹分布。
>>> import numpy as np
>>> from scipy.stats.sampling import SimpleRatioUniforms
假设我们有正态分布
>>> class StdNorm:
... def pdf(self, x):
... return np.exp(-0.5 * x**2)
请注意,PDF 未积分到 1。我们可以传递 PDF 初始化生成器期间 PDF 下方的确切面积,或者传递 PDF 下方的确切面积的上限。此外,建议传递分布的模式以加快设置速度
>>> urng = np.random.default_rng()
>>> dist = StdNorm()
>>> rng = SimpleRatioUniforms(dist, mode=0,
... pdf_area=np.sqrt(2*np.pi),
... random_state=urng)
现在,我们可以使用 rvs 方法从分布中生成样本
>>> rvs = rng.rvs(10)
如果模式下的 CDF 可用,则可以设置它以提高 rvs 的性能
>>> from scipy.stats import norm
>>> rng = SimpleRatioUniforms(dist, mode=0,
... pdf_area=np.sqrt(2*np.pi),
... cdf_at_mode=norm.cdf(0),
... random_state=urng)
>>> rvs = rng.rvs(1000)
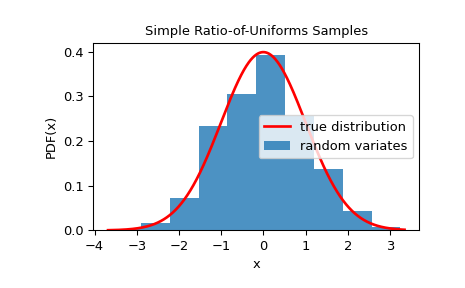
我们可以通过可视化其直方图来检查样本是否来自给定分布
>>> from scipy.stats.sampling import SimpleRatioUniforms
>>> from scipy.stats import norm
>>> import matplotlib.pyplot as plt
>>> class StdNorm:
... def pdf(self, x):
... return np.exp(-0.5 * x**2)
...
>>> urng = np.random.default_rng()
>>> dist = StdNorm()
>>> rng = SimpleRatioUniforms(dist, mode=0,
... pdf_area=np.sqrt(2*np.pi),
... cdf_at_mode=norm.cdf(0),
... random_state=urng)
>>> rvs = rng.rvs(1000)
>>> x = np.linspace(rvs.min()-0.1, rvs.max()+0.1, 1000)
>>> fx = 1/np.sqrt(2*np.pi) * dist.pdf(x)
>>> fig, ax = plt.subplots()
>>> ax.plot(x, fx, 'r-', lw=2, label='true distribution')
>>> ax.hist(rvs, bins=10, density=True, alpha=0.8, label='random variates')
>>> ax.set_xlabel('x')
>>> ax.set_ylabel('PDF(x)')
>>> ax.set_title('Simple Ratio-of-Uniforms Samples')
>>> ax.legend()
>>> plt.show()
该方法的主要优点是设置速度快。如果需要重复生成具有不同形状参数的分布的小到中等样本,这将是有益的。在这种情况下,sampling.NumericalInverseHermite 或 sampling.NumericalInversePolynomial 的设置步骤将导致性能不佳。例如,假设我们有兴趣为 Gamma 分布生成 100 个样本,其中 1000 个不同的形状参数由 np.arange(1.5, 5, 1000) 给出。
>>> import math
>>> class GammaDist:
... def __init__(self, p):
... self.p = p
... def pdf(self, x):
... return x**(self.p-1) * np.exp(-x)
...
>>> urng = np.random.default_rng()
>>> p = np.arange(1.5, 5, 1000)
>>> res = np.empty((1000, 100))
>>> for i in range(1000):
... dist = GammaDist(p[i])
... rng = SimpleRatioUniforms(dist, mode=p[i]-1,
... pdf_area=math.gamma(p[i]),
... random_state=urng)
... with np.testing.suppress_warnings() as sup:
... sup.filter(RuntimeWarning, "invalid value encountered in double_scalars")
... sup.filter(RuntimeWarning, "overflow encountered in exp")
... res[i] = rng.rvs(100)